A reação em cadeia da polimerase (PCR) se tornou nos dias atuais uma das tecnologias mais utilizadas e bem vista na biologia molecular, se você deseja amplificar/copiar milhões de vezes sequências especificas dentro de um DNA ou molde de cDNA,através do uso de oligonucleotídeos específicos de sequência, DNA polimerase HotStart e ciclos de temperatura, esta é técnica ideal!
Na PCR tradicional a reação acontece em termociclador comum, e a quantificação da sequência amplificada são realizadas no final da reação após o último ciclo de PCR, e a análise é feita através da eletroforese horizontal em gel de agarose (Confira mais no nosso blog), seguida da análise de imagem através de um transluminador UV. Na PCR quantitativa em tempo real (qPCR), o produto da PCR é medido em cada ciclo, sendo possível monitorar as reações durante a fase de amplificação exponencial da reação no termociclador para qPCR, os usuários podem determinar a quantidade inicial do alvo com grande precisão.
A PCR em tempo real (qPCR) é uma variação da PCR tradicional, utilizada para quantificação de DNA e RNA usando sequências especificas de primers ou sondas (probes), e o número de cópias da sequência desejada de DNA ou RNA é determinada. A quantificação do produto amplificado de PCR é mensurada através de ciclos de PCR usando sondas fluorescentes ou corantes fluorescentes de ligação a DNA e instrumentos de PCR em tempo real que medem a fluorescência enquanto realizam o ciclo térmico necessário para a reação. A PCR acontece em 3 etapas, que correm em torno de 40-45 ciclos: Denaturação, processo para separar a fita dupla de DNA em fita simples; Anelamento, durante esta etapa a sequência complementar é hibridizada através da temperatura ideal, mensurando a temperatura de melting, ideal para os primers; e Extensão, a temperatura ideal para a enzima Taq DNA polimerase, assim ocorre a extensão dos primers, em uma média de 100 pares de base/segundo. A reação de qPCR pode acontecer em 1 ou 2 etapas, confira:
Two-Step qPCR (2 etapas)
A reação de PCR de transcriptase reversa (RT) pode ser feita em duas etapas, começa com a transcrição reversa do RNA total ou poli (A) RNA em cDNA usando a enzima transcriptase reversa (RT). Esta reação de síntese de cDNA de primeira fita pode ser iniciada usando primers aleatórios. A temperatura usada para a síntese de cDNA depende da enzima RT escolhida. Após a transcrição reversa, aproximadamente 10% do cDNA é transferido para um outro tubo para a reação de PCR em tempo real.

One step qRT-PCR (1 etapa)
Para otimização de tempo, a qRT-PCR one step combina a reação de síntese do cDNA com a reação de PCR em tempo real no mesmo tubo, simplificando a configuração da reação e reduzindo a possibilidade de contaminação.
Componentes e sua importância
DNA polimerase
O desempenho do PCR está frequentemente relacionado a enzima termoestável Taq DNA polimerase, então a seleção da enzima é crucial para o sucesso. Um dos principais fatores que afetam a especificidade da PCR é o fato de que a enzima tem atividade residual em baixas temperaturas, ou seja, a utilização de uma enzima resistente a altas temperaturas é uma boa escolha, como as enzimas HotStart. Os primers podem fazer o pareamento de forma não específica, permitindo que a polimerase sintetize produto não específico. Usando uma enzima HotStart garante que a DNA polimerase não está ativa durante a etapa inicial de desnaturação do DNA.
Transcriptase reversa
A transcriptase reversa (RT) é tão importante quanto a enzima Taq DNA polimerase. É importante selecionar uma RT que não só forneça altos rendimentos de comprimento total cDNA, mas também tenha boa atividade em altas temperaturas. Na reação one step da RT-qPCR, a RT se torna inativa em elevadas temperaturas, que permitem que você use primers específicos com alta temperatura de melting, aumentando a especificidade e reduzindo ruídos.
dNTPs
É uma boa ideia comprar os dNTPs e os DNA polimerase termoestável do mesmo fornecedor, como não é incomum ver uma perda de sensibilidade de um ciclo de limiar completo (Ct) em experimentos que empregam esses reagentes de fornecedores diferentes.

Concentração de magnésio
Em PCR em tempo real, cloreto de magnésio é normalmente usada em uma concentração final de 3 mM. Essa concentração funciona bem para a maioria dos alvos; Contudo, a concentração ideal de magnésio pode variar entre 3 e 6 mM.
Amostra de DNA/RNA
Use de 10 a 1.000 cópias de amostras de DNA para cada reação de qPCR, é equivalente a aproximadamente 100 pg a 1 μg de DNA genômico, ou cDNA gerado a partir de 1 pg a 100 ng de RNA total. O excesso de amostra também pode trazer níveis mais altos de contaminantes que podem reduzir a eficiência da reação. Dependendo da especificidade dos primers de PCR para cDNA em vez de DNA genômico, pode ser importante tratar modelos de RNA para reduzir a chance de que eles contenham contaminação de DNA genômico. O RNA total normalmente funciona bem em qRT-PCR; isolamento de mRNA normalmente não é necessário, embora pode melhorar o rendimento de cDNAs específicos.
A detecção fluorescente da reação qPCR
Existem diversas formas da detecção fluorescente no PCR em tempo real, as mais conhecidas são os ensaios de 5’ nuclease, como TaqMan, e ensaios baseados em corante SYBR Green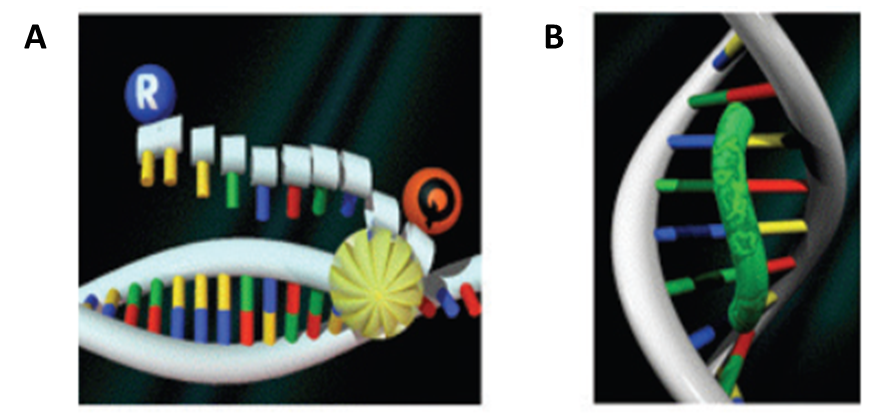
Figura 1. Representação da marcação por TaqMan (A) e SYBR Green (B)
Durante a PCR, os primers e a sonda se ligam ao alvo. A DNA polimerase estende o primer adiante da sonda. Se a sonda estiver ligada à sequência de destino correta, a atividade de 5’ nuclease da polimerase cliva a sonda, liberando um fragmento contendo o corante repórter, ocorrendo a fluorescência da amostra.
As sondas podem ser divididas em dois tipos: MGB e não-MGB. No início do desenvolvimento de PCR em tempo real, testes extensivos revelaram que as sondas exigiam uma temperatura de anelamento significativamente mais alta do que primers comuns de PCR para permitir que ocorra a clivagem, portanto as sondas são mais longas que os primers. Contudo, para muitas aplicações envolvendo alta complexidade genética, tais como expressão de genes eucarióticos e nucleotídeo único polimorfismos (SNPs), um maior grau de especificidade a sanda é recomendada. O corante SYBR® Green I é um corante fluorescente de ligação ao. A excitação do corante SYBR® Green ligado ao DNA produz um sinal fluorescente muito mais forte em comparação com o não ligado corante. Uma reação de PCR corado pelo SYBR® Green normalmente consiste em dois primers. Em condições ideais, um SYBR® Green ensaio segue um padrão de amplificação semelhante ao de sonda. Se o alvo estivesse presente na amostra, o produto de PCR se for amplificado o suficiente, ocorrerá a fluorescência pelo corante.
Outro destaque dentro da qPCR, é o corante passivo de referência, geralmente é utilizado nos termocicladores de PCR em tempo real, esse tipo de normalização corrigi a variação de poço/poço que podem alterar as concentrações ou volume da reação, alterando a leitura da placa. A maioria dos instrumentos de qPCR utilizam o corante de referência passivo ROX, pois ele não afeta a reação e seu sinal de fluorescência é distinto dos corantes fluorescentes utilizados (reporter ou quencher). O uso de um corante de referência passivo é uma ferramenta eficaz para a normalização do repórter fluorescente sinalizar sem modificar a análise padrão do instrumento parâmetros.
• Normalizar para flutuações não relacionadas a PCR em fluorescência (por exemplo, causada pela variação na pipetagem)
• Normalizar para flutuações na fluorescência resultante do “ruído” da máquina
• Compensar as variações na excitação do instrumento e detecção

 Notícias
Notícias


